|
Molecola del mese di agosto 2000
Gli enzimi di restrizione sono enzimi batterici
che tagliano il DNA virale e ora sono usati anche dall'ingegneria genetica
 I
batteri si difendono I
batteri si difendono
I batteri sono continuamente attaccati dai virus batteriofagi, come il
batteriofago phiX174. Per proteggere sè stessi molti batteri hanno sviluppato
un metodo per tagliare le molecole di DNA estraneo, come quella di un
fago che li sta infettando. Questi batteri sintetizzano un'endonucleasi,
un enzima che taglia il DNA, che rimane nel citoplasma del batterio pronto
a colpire il DNA del fago. Queste endonucleasi sono chiamate enzimi di
restrizione perchè limitano l'infezione dei batteriofagi (restriction
significa limitazione in inglese).
Forbici molecolari
Ogni tipo di enzima di restrizione riconosce una particolare sequenza
di DNA e la taglia con precisione in un punto. Per esempio, l'enzima mostrato
qui a lato, EcoRI (eco erre uno), riconosce la sequenza GAATTC
e la taglia tra G e A. Naturalmente le endonucleasi libere, nel citoplasma
di un batterio, possono essere pericolose, così i batteri proteggono il
proprio DNA modificando con dei gruppi metilici le sequenze bersaglio
dei propri enzimi di restrizione. Gruppi metilici vengono aggiunti
alle basi azotate adenina o citosina (a seconda del tipo di batterio)
nella fenditura maggiore del DNA. I gruppi metilici impediscono agli enzimi
di restrizione di legarsi, ma non ostacolano la normale attività
di traduzione e duplicazione del DNA. Un batteriofago che sta attaccando
il batterio non possiede questi gruppi protettori metilici nel suo DNA
e viene distrutto. Ogni batterio ha un suo insieme di enzimi di restrizione
che tagliano sequenze specifiche del DNA, accoppiati con specifici enzimi
metil-trasferasi che proteggono le stesse sequenze nel genoma del
batterio.
Terminazioni appiccicose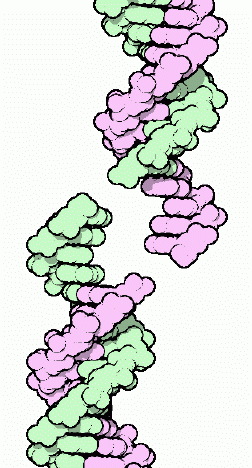
Il rapido sviluppo delle biotecnologie è stato reso possibile dalle scoperta
degli enzimi di restrizione nei primi anni 1950. Questi tagliano il DNA
in punti precisi. Un altro enzima, DNA ligasi, può essere usato per ricomporre
i frammenti nell'ordine desiderato. Questi due enzimi, insieme, permettono
ai ricercatori di costruire nuove sequenze genomiche. Per esempio, sono
stati creati batteri che sintetizzano l'insulina e l'ormone della crescita,
e sono stati aggiunti geni per la resistenza a certe malattie alle piante
coltivate.
Gli enzimi di restrizione riconoscono una sequenza palindroma di
DNA, come quella di EcoRI, GAATTC, formata da basi simmetricamente complementari.
La sequenza GAATTC è detta palindroma perchè
può essere letta da sinistra a destra sulla catena superiore e
da destra a sinistra su quella inferiore.
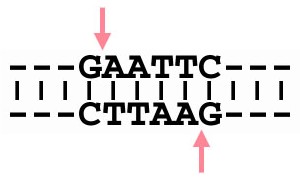
Una proprietà interessante degli enzimi di restrizione
semplifica il lavoro di taglio e assemblaggio. Quando l'enzima, con un
taglio asimmetrico, taglia le catene tra G e A, lascia due frammenti che
terminano entrambi con un segmento a singola catena:

Questi due frammenti di DNA possiedono terminazioni
appiccicose perchè le basi che si formano sui segmenti sporgenti sono
complementari e quindi tendono a riunire insieme i due frammenti anche
se la sequenza è tagliata. Le terminazioni appiccicose sono una componente
importante dell'ingegneria genetica, perchè permettono ai ricercatori
di unire tra loro anche frammenti di DNA di diversa provenienza, purchè
ottenuti con lo stesso enzima di restrizione.
Esplorando la struttura
Non tutti gli enzimi di restrizione producono terminazioni appiccicose,
alcuni generano terminazioni piatte. EcoRV (eco erre cinque),
per esempio, un altro enzima preso da Escherichia coli, riconosce la
sequenza GATATC e opera un taglio simmetrico GAT-ATC producendo terminazioni
piatte. Sono disponibili strutture di questo enzima che lo riprendono
sia prima che dopo la reazione di taglio del DNA.
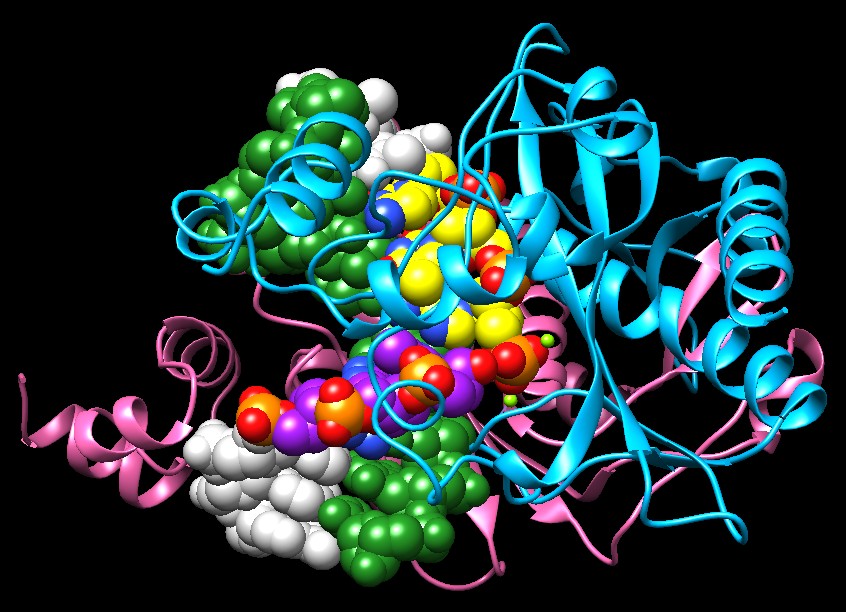
La struttura qui sotto (file PDB 1rva) mostra l'enzima EcoRV legato ad un corto frammento
di DNA non ancora tagliato, le cui catene sono colorate una in verde
e l'altra in bianco.
Per mettere in evidenza il punto dove avverrà il taglio nella
catena bianca, i sei nucleotidi della sequenza GATATC in questa catena
sono stati colorati con due colori diversi: GAT con i carboni in viola
e i successivi ATC con i carboni in giallo. Il taglio avviene quindi
al confine tra i nucleotidi viola e gialli: il gruppo fosfato in 5'
dell'adenina (gialla), evidenziato da un asterisco, verrà staccato
dall'ossigeno 2' del ribosio della timina (viola) che si trova subito
sotto.
. . . . . . . . .
La figura seguente permette di vedere meglio gli atomi coinvolti nella
reazione. Il taglio avverrà tra il fosfato in 5' dell'adenosina
gialla, evidenziato da un asterisco, e l'OH 2' del ribosio viola della
timina.
. . . . . . . 
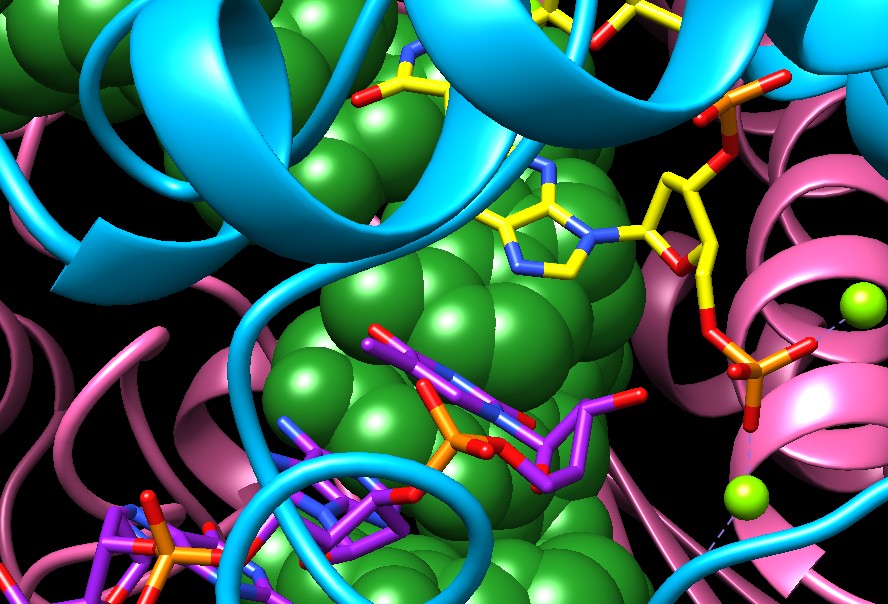
La figura qui sotto (file PDB 1rvc) mostra l'enzima EcoRV dopo che il DNA è stato
tagliato. Qui osserviamo il taglio solo sulla catena bianca all'interno
dei sei nucleotidi colorati di viola e giallo. Un taglio identico è
avvenuto anche sulla catena verde. L'idrolisi del legame estere fosforico
ha liberato l'OH 2' della timina dal gruppo fosforico dell'adenina gialla.
Nella figura questi due ossigeni si vedono uno vicino all'altro. Alla
reazione hanno contribuito anche i due ioni magnesio Mg2+
visibili vicino al fosfato come due sferette verdi.
. . . . . . . . . 
La figura che segue permette di vedere meglio gli atomi coinvolti nella
reazione. Il taglio che ha separato in due frammenti la catena del DNA
è avvenuto tra il fosfato in 5' dell'adenosina gialla (sulla
destra) e l'OH 2' del ribosio viola della timina (al centro).
. . . . . . 
|
|
|
 I
batteri si difendono
I
batteri si difendono






