|
Molecola del mese di marzo 2001
L'RNA transfer traduce il linguaggio di
nucleotidi dell'RNA messaggero (copiato dal DNA) , nel linguaggio di amminoacidi
delle proteine
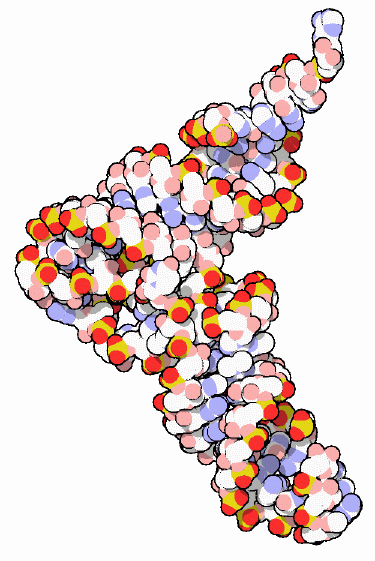 Introduzione Introduzione
Da quando è stato scoperto il processo di sintesi
delle proteine diretto dal DNA, scienziati e filosofi hanno cercato una
relazione tra le triplette di acidi nucleici, i codoni, e la natura chimica
degli amminoacidi. Questi tentativi sono stati tutti infruttuosi, ma il
tema continua occasionalmente a stimolare riflessioni per le possibili
implicazioni con l'origine della vita. In realtà, non sembra che
ci sia una relazione specifica tra i codoni e gli amminoacidi in quanto
tali. Invece, la corrispondenza è realizzata dall'RNA transfer
(tRNA), una specie di Stele di Rosetta della biochimica, che traduce
il linguaggio di nucleotidi dell'RNA messaggero (copiato dal DNA) nel
linguaggio di amminoacidi delle proteine. Questa traduzione è fisica
e diretta: a un capo di ogni tRNA c'è un anticodone che riconosce
il codone del codice genetico, all'altro capo si trova legato il giusto
amminoacido corrispondente a quel codice.
Alta fedeltà
Errori nella sintesi delle proteine possono accadere
ad entrambi i capi del tRNA. All'estremità 3' del tRNA deve essere
legato il giusto amminoacido, pronto per essere aggiunto alla
catena della proteina in crescita. Un gruppo di enzimi, chiamati amminoacil
tRNA sintetasi (mdm 4/2001) è responsabile di questa
operazione. Questi enzimi mediamente commettono un errore ogni diecimila
molecole di tRNA a cui hanno legato un amminoacido.
All'altra estremità del tRNA l'anticodone si deve accoppiare
con il suo codone complementare. Una volta su cinquecento, però,
può accadere che l'anticodone si accoppi in modo errato.
Oppure, dato che ogni codone è lungo tre nucleotidi, l'anticodone
si può accoppiare in una posizione sfasata, invece di
sovrapporsi alla giusta tripletta. Questo porta errori in tutta la parte
restante della proteina, perchè tutti i successivi tRNA si allineano
in posizione sfasata in conseguenza del primo spostamento. Fortunatamente,
quando le sequenze genetiche vengono lette in modo sfasato, si possono
formare facilmente codoni di STOP, così la sintesi della
proteina è interrotta dopo che sono state aggiunte solo poche
decine di amminoacidi in più.
Ricavare il massimo dagli errori
L'evoluzione biologica è straordinaria nella
sua abilità di trarre profitto anche dagli errori. Se c'è
un modo per trasformare un problema in un vantaggio, sicuramente il
processo di selezione naturale lo troverà. Gli errori nella sintesi
delle proteine non fanno eccezione. Tutti e due gli accoppiamenti errati,
quello codone-anticodone e quello di sfasamento di lettura, hanno un
particolare ruolo funzionale in certi organismi.
Spesso, vengono usati codoni diversi come segnali di INIZIO al posto
di AUG. Questi codoni possono essere GUG, UUG, o AUU, ma tutti usano
lo stesso tRNA, quello della metionina che normalmente riconosce il
codone AUG. Per poter sintetizzare queste proteine, il tRNA della metionina
si deve appaiare con questi codoni erronei.
Lo sfasamento della lettura è essenziale per il ciclo di vita
del virus HIV. Quando sintetizzano la lunga poliproteina che contiene
tutte le proteine che si trovano nel virus, i ribosomi fanno un errore
di accoppiamento nel 5% dei casi, allineando in modo errato un tRNA
e provocando uno sfasamento della lettura. Questo impedisce al ribosoma
di incontrare il normale codone di STOP e così viene prodotta
una proteina molto più lunga. Questi errori occasionali sono
fondamentali per la vita del virus HIV, perché solo le proteine
più lunghe contengono gli enzimi che trascrivono il genoma virale.
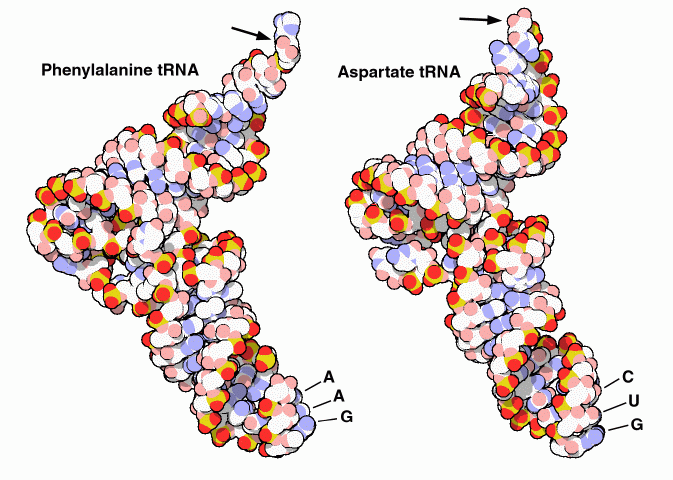 RNA
transfer RNA
transfer
Le molecole di RNA transfer sono composte da una
corta catena di RNA, lunga 70-90 nucleotidi, piegata a forma di trifoglio.
Qui a fianco sono mostrate due molecole diverse, fenilalanina tRNA (file
PDB 4tna) e acido aspartico tRNA (file
PDB 2tra).
L'estremità 3' delle catene di RNA si trova nella parte alta
della figura dove le strutture si fanno più sottili. Qui, indicato
da una freccia, si trova l'amminoacido legato all'OH in posizione
3' sul nucleotide terminale.
Il centro della catena forma la porzione arrotondata in basso, dove
affiorano i tre nucleotidi che formano l'anticodone. Gli altri
due lobi del trifoglio sono intrecciati insieme nella zona a gomito
e danno una struttura definita a tutta la molecola. Le quattro basi
azotate dell'RNA (adenina, uracile, guanina e citosina) non riescono
da sole a dare una struttura robusta alla molecola anche perché
molte hanno una struttura modificata.
Per vedere due esempi di basi modificate, osservate la base numero 37,
quella vicina all'anticodone, nel metionina tRNA (il tRNA che dà
inizio alla sintesi, file PDB 1yfg) o
nel fenilalanina tRNA (file PDB 4tna e
6tna).
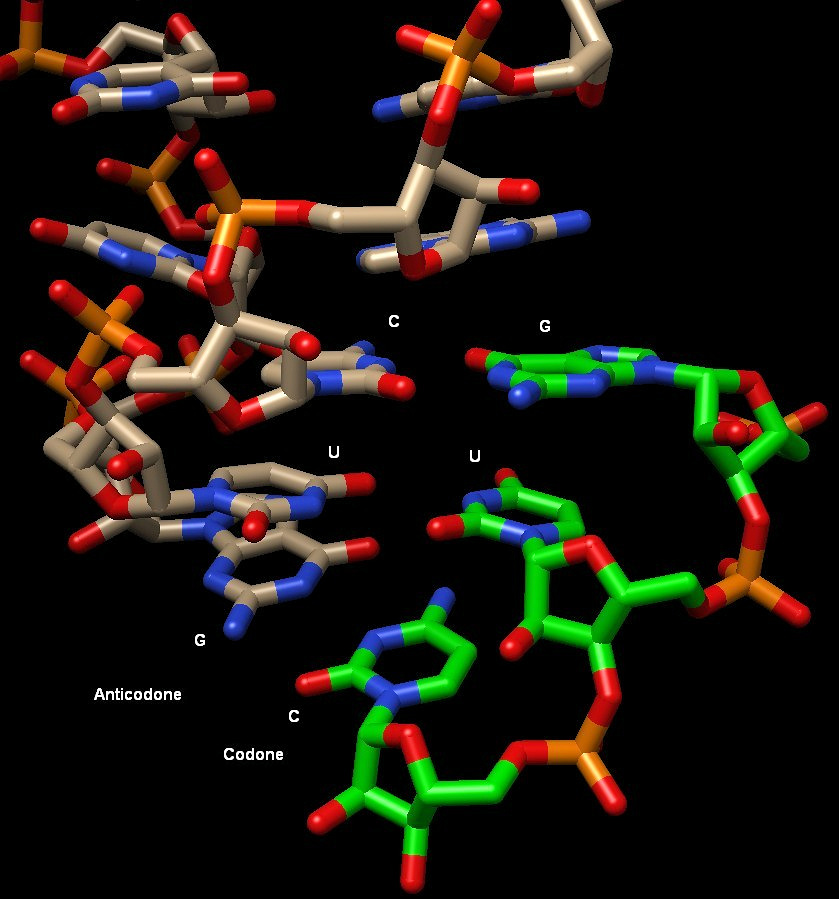
Esplorando la struttura
Naturalmente, la prima cosa che esaminiamo nella
struttura del tRNA è l'anticodone. Nella figura qui sotto
si può vedere un acido aspartico tRNA (file PDB 2tra).
Quando è stato fatto cristallizzare per sottoporlo all'analisi
ai raggi X, questo tRNA ha formato un dimero nel quale gli anticodoni
di due molecole si sono legati insieme. Il segmento dalla seconda molecola,
mostrato nella parte bassa della figura (con i carboni colorati in verde),
ci può dare un'idea di come il codone dell'RNA messaggero si
legherebbe all'anticodone del tRNA. L'anticodone in questa struttura
è composto (dal basso verso l'alto) da guanina, uracile, e citosina.
La prima coppia dal basso (G-C) e la terza (C-G) sono perfettamente
accoppiate, mentre la coppia centrale è composta da due basi
azotate piccole (U-U) che non si possono accoppiare, ma almeno, dato
restano lontane tra di loro, non interferiscono.
. . . . . . . . . 
Nel fenilalanina tRNA (file PDB 4tna)
vi sono alcune tiplette di nucleotidi, come quella mostrata qui
sotto. Si tratta di una strana interazione che coinvolge tre basi ed
è simile a quella che si trova negli aptameri
di RNA fluorescenti (mdm 1/2019). Citosina e guanina si accoppiano
come un normale paio di basi, come nel DNA, ma qui, nell'intreccio della
catena, si avvicina un'altra guanina, modificata con un gruppo metilico
(piccola sfera), che forma un'insolita interazione legandosi lateralmente
al paio di basi. I legami idrogeno che si formano sono indicati con
sottili fili azzurri.
. . . . . . . . . .
. . . . . . 
 Bibliografia Bibliografia
John G. Arnez and Dino Moras (1999) Transfer
RNA. In "Oxford Handbook of Nucleic Acid Structure"
(Stephen Neidle, Editor) Oxford University Press. Pages 603-651.
Alexander Rich and Sung Hou Kim (1978) The Three-dimensional
Structure of Transfer RNA. Scientific American 238 (No.
1), 52-62.
Jack Parker (1989) Errors and Alternatives in Reading the Universal
Genetic Code. Microbiological Reviews 53, 273-298.
|
|
|
 Introduzione
Introduzione
 RNA
transfer
RNA
transfer 
