|
Molecola del Mese di Ottobre
2010
Speciali sequenze nell'mRNA sono interruttori
che possono fermare la sintesi proteica se si legano ad opportune molecole
regolatorie
 Introduzione Introduzione
Perché usare più molecole quando ne
potrebbe bastare una sola? Nelle nostre cellule la sintesi proteica è
controllata da migliaia di proteine regolatrici che collaborano per decidere
quando una determinata proteina deve essere sintetizzata. I batteri, invece,
che sono maestri di economia, hanno trovato il modo, in alcuni casi, di
indurre l'RNA messaggero a controllarsi da solo senza l'aiuto di proteine.
Autocontrollo
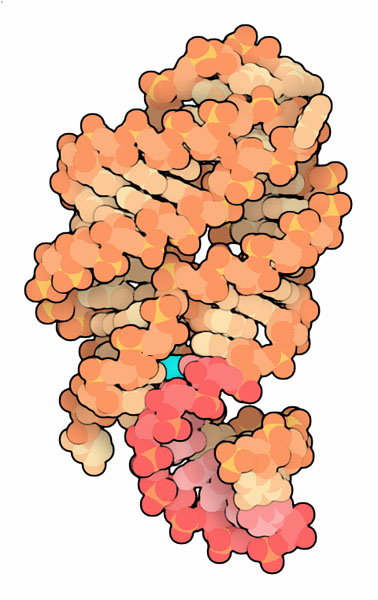
I riboswitch sono elementi regolatori inseriti direttamente
nell'RNA messaggero. Per esempio il riboswitch mostrato qui a fianco
(file PDB 1u8d) sente i livelli delle
basi puriniche legandosi saldamente a guanina, ipoxantina, e xantina.
Questo riboswitch è parte dell'RNA messaggero che codifica per
enzimi che trasportano e metabolizzano purine. Così, quando ci
sono molte basi puriniche, queste si legano al riboswitch e rallentano
la produzione di quelle proteine che in quel momento non servono.
Girare l'interruttore
I riboswitch possono agire in due modi.
Primo, quando il riboswitch è legato ad una molecola di legando,
assume una forma ripiegata, così la cellula può sentire
il livello di quel legando osservando la forma del riboswitch.
Secondo, una parte del riboswitch controlla
l'espressione dell'RNA messaggero. Quando il riboswitch ha al suo interno
il legando, si dispone in una conformazione ripiegata che significa
interruttore spento. Quando invece il legando non è disponibile,
il riboswitch si dispone in una conformazione rilassata che significa
interruttore acceso e controlla la trascrizione dell'RNA messaggero
e quindi la sintesi proteica, in alcuni casi aumentandola in altri fermandola.
Riboswitch dovunque
Nei batteri sono stati trovati molti riboswitch
e ne sono stati individuati alcuni anche nelle piante e nei funghi.
Sono in grado di sentire il livello di molti tipi di molecole, non solo
di basi azotate, ma anche di amminoacidi, vitamine e coenzimi.
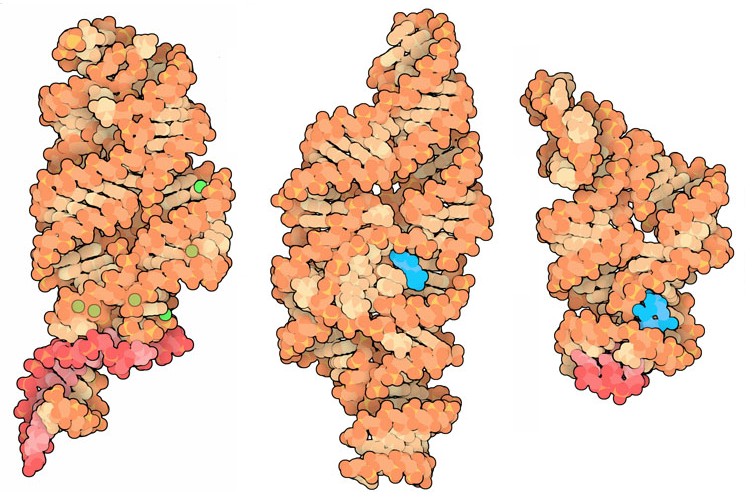
Qui sotto sono illustrati tre esempi significativi.
Sulla sinistra è mostrato un riboswitch che sente i livelli
di ioni magnesio (verdi) (file PDB 2qbz).
Al centro vi è un riboswitch che sente i livelli di glucosammina-6-fosfato
(azzurra), e che poi si trasforma in un ribozima che taglia se stesso
(file PDB 2z75).
A destra vi è un riboswitch che sente c-di-GMP
(guanilato dimerico ciclico, azzurro) (file PDB 3irw),
un nucleotide che è usato come messaggero, un ruolo simile a
quello svolto da cAMP (AMP ciclico) nelle nostre cellule.
. . . . . . . . .. . . . . . . 
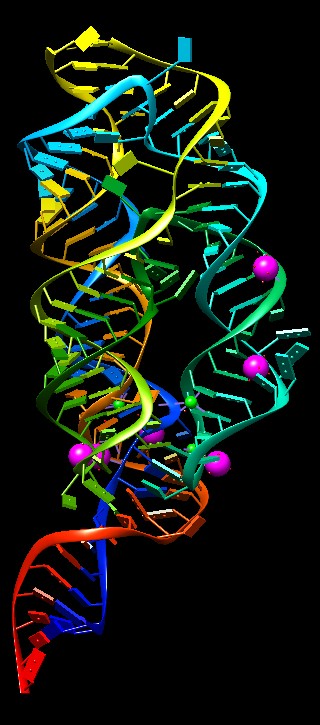
Gli stessi 3 ribozimi sono mostrati qui sotto rappresentati con Chimera.

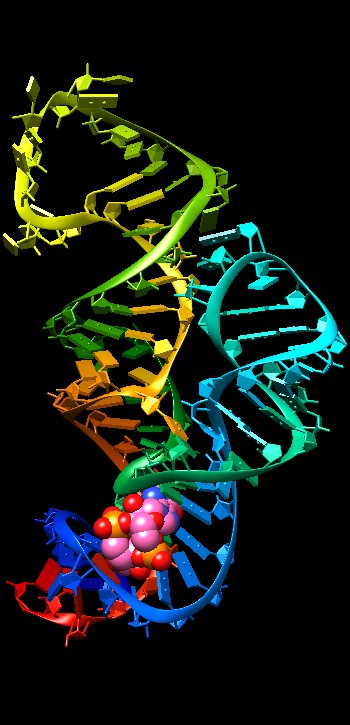
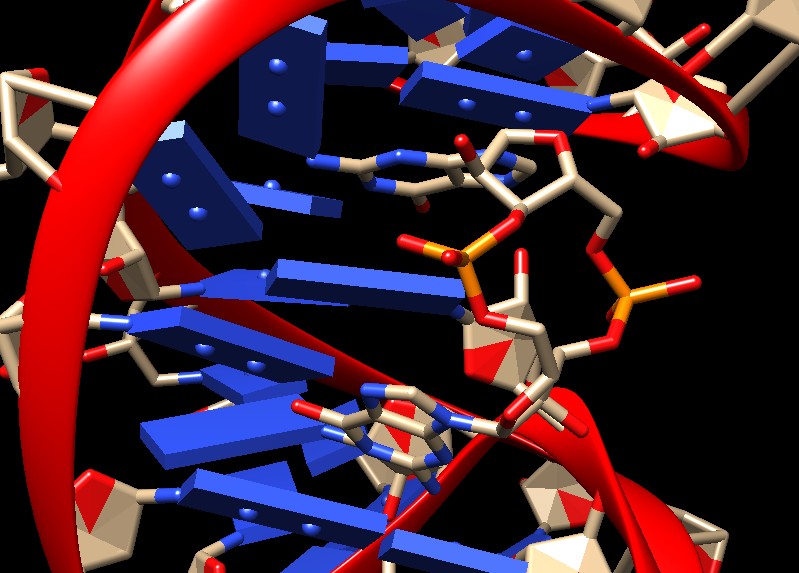
La molecola di c-di-GMP, guanilato dimerico ciclico, del terzo riboswitch
è mostrata in maggior dettaglio qui sotto.
Le due guanine del c-di-GMP si appaiano con le basi azotate complementari
nel punto dove la catena dell'RNA del riboswitch si piega e perde per
un breve tratto la struttura a doppia elica lasciando scoperte alcune
basi azotate (file PDB 3irw).
. . . . . . . . . . . . 
Esplorando la struttura
I riboswitch sono notevolmente specifici per il
proprio legando e lo catturano con una forza e una specificità
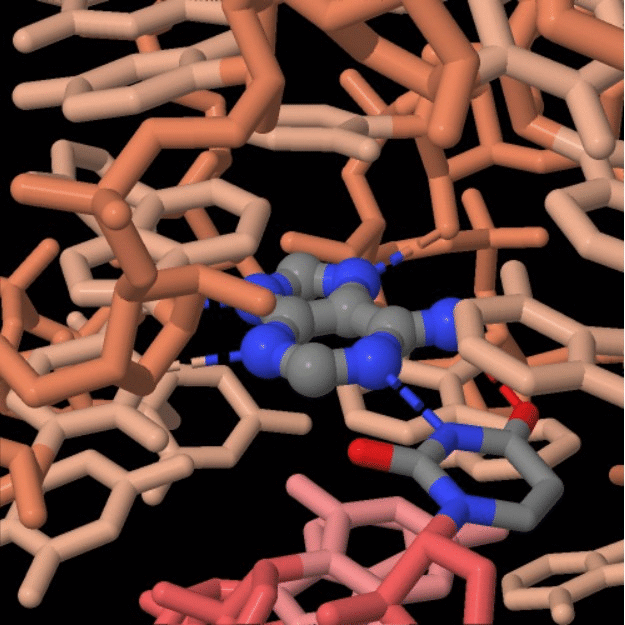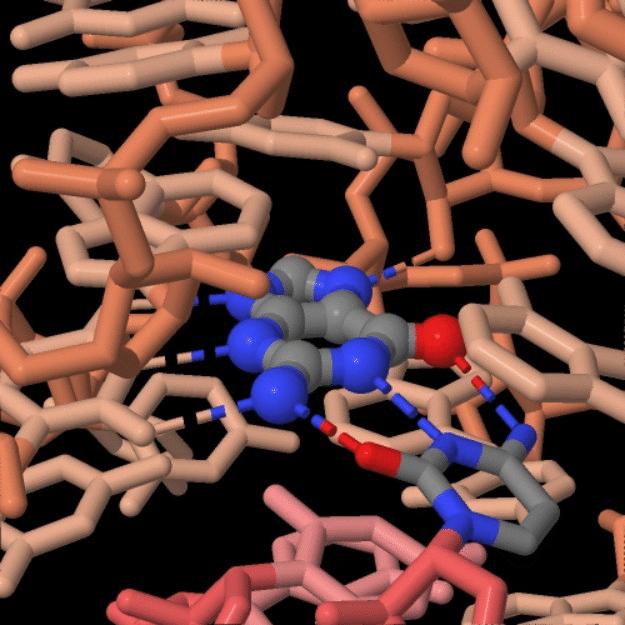
simili a quella degli enzimi proteici. Nell'immagine qui sotto, potete
vedere alternativamente due riboswitch simili (file PDB 1y26
e 1y27), il primo ha come legando l'adenina
e l'altro la guanina (molecole con sfere al centro dell'immagine).
I due riboswitch hanno una sequenza di nucleotidi quasi identica e sono
ripiegati quasi nello stesso modo. La chiave della loro specificità
è nel nucleotide in posizione 74 (anello colorato sulla
sinistra). Questo nucleotide forma una tipica coppia di basi secondo
Watson-Crick con il legando (molecola al centro con sfere) così
può legare in modo selettivo adenina (nel primo riboswitch)
con due legami idrogeno (mostrati con tratteggi) o guanina (nel
secondo) con tre legami idrogeno.
I legandi adenina e guanina sono mostrati al centro con l'opzione balls-and-sticks
a colori, mentre il nucleotide 74 è mostrato sulla sinistra con
una struttura tubolare colorata.
Nel primo riboswitch il nucleotide 74 è uracile e forma
2 legami idrogeno con adenina.
Nel secondo riboswitch il nucleotide 74 è citosina e forma
3 legami idrogeno con guanina.
. . . . . .. .. .
. . . . . . . .. . . . . . 
Nell'immagine qui sotto è mostrato l'appaiamento di basi adenina-uracile
e guanina-citosina visto da una diversa angolazione.
.. . . . . . . . . . . . . . . . . . . ... . 
Spunti per ulteriori esplorazioni
1. Negli archivi PDB ci sono le strutture
di molti altri riboswitch. Ne sapete trovare qualche esempio?
2. I riboswitch riconoscono il loro legando usando solo le quattro
basi dell'RNA. Usate il Ligand Explorer nel sito PDB per scoprire come
altri riboswitch riconoscono legandi diversi.
Cominciate, per esempio, cliccando
qui per esaminare il riboswitch che lega S-adenosilmetionina.
 Bibliografia Bibliografia
A. Serganov (2009) The long and short of
riboswitches. Current Opinion in Structural Biology 19,
251-259.
M. D. Dambach, W. C. Winkler (2009) Expanding roles for metabolite-sensing
regulatory RNAs. Current Opinion in Microbiology 12, 161-169.
R. K. Montange, R. T. Batey (2008) Riboswitches: Emerging themes
in RNA structure and function. Annual Review of Biophysics 37,
117-133.
|
|
|
 Introduzione
Introduzione






