|
|
Aptameri di RNA fluorescenti |
||||||
|
Molecola del Mese di Gennaio 2019 Gli aptameri di RNA vengono ingegnerizzati per seguire le molecole all'interno di cellule viventi  Introduzione IntroduzioneGli scienziati sono sempre alla ricerca di nuovi strumenti che consentano di esplorare le cellule con un dettaglio sempre maggiore. La proteina verde fluorescente GFP (mdm 6-2003) è stata una dei primi strumenti di questo tipo e ha permesso di esplorare la cellula in modi completamente nuovi, rendendo visibili specifiche proteine per osservarne il comportamento all'interno di una cellula vivente. Recentemente gli scienziati hanno sviluppato un nuovo strumento che permette di osservare nello stesso modo l'RNA. L'RNA, di per sé, non è fluorescente così è stato costruito ex-novo un breve tratto di RNA in grado di legarsi ad un fluoroforo (una piccola molecola fluorescente) formando un complesso RNA-fluoroforo ancora più fluorescente. A questo punto, con tecniche di ingegneria genetica, questo RNA è stato inserito in un RNA naturale, come quello di un ribosoma. Quando si somministra il fluoroforo alla cellula, questo si lega al ribosoma modificato che così diventa fluorescente e permette di seguirne i movimenti. Far evolvere gli aptameri Le molecole di RNA che legano il fluoroforo non esistevano in natura, per ottenerle gli scienziati hanno sviluppato una tecnica innovativa chiamata SELEX: systematic evolution of ligands by exponential enrichment. Questa procedura comincia mescolando il fluoroforo con molte piccole catene di RNA casuali e poi isolando le catene che hanno legato il fluoroforo. Queste catene vengono poi modificate in più modi casuali e le migliori, quelle che legano meglio il fluoroforo, vengono ancora isolate. Dopo molti cicli di modifica e selezione si ottiene un aptamero (molecola che è in grado di legarsi ad una piccola molecola) che si lega efficacemente con il suo fluoroforo e ne aumenta la fluorescenza. L'aptamero mostrato qui a fianco è soprannominato "Spinacio" perchè emette una luce fluorescete verde quando è legato al suo fluoroforo (in verde nella figura qui a lato). Questo fluoroforo (difluoro-idrossibenzil-imidazolo DFHBI.pdb) è simile a quello presente nella proteina verde fluorescente, ed è mostrato nella figura qui sotto. Sull'anello di destra sono presenti due atomi di fluoro (verdi). 
Sfide strutturali L'aptamero Spinacio ha la struttura di una lunga forcina, con al centro una sezione ripiegata in modo più complesso che circonda il fluoroforo. Dato che l'RNA è difficile da cristallizzare, per determinare la struttura di questo aptamero si sono dovuti usare vari artifici. L'aptamero Spinacio del file PDB 4kzd (qui sopra a destra) ha uno degli avvolgimenti modificato in modo da legare un anticorpo che ha aiutato a formare cristalli più stabili. L'aptamero Spinacio del file PDB 4ts2 (mostrato più sotto in esplorando la struttura) è stato privato di qull'avvolgimento in modo da consentire alle molecole di RNA di impaccarsi più strettamente per formare cristalli stabili. 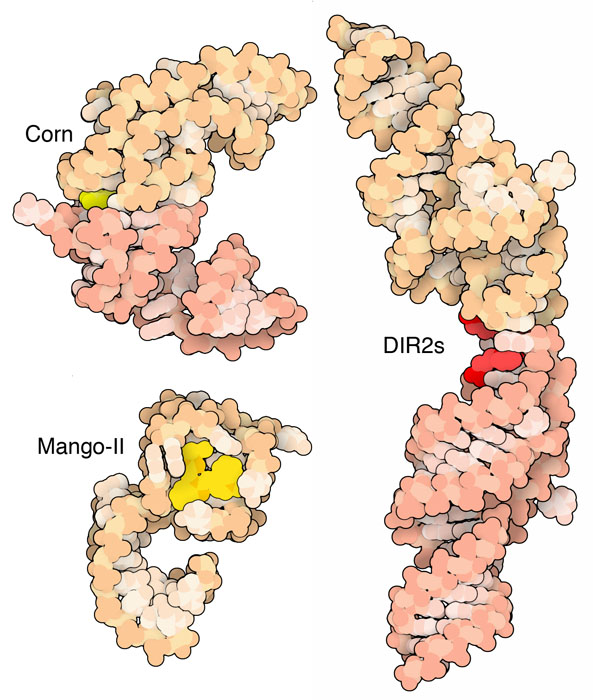 Aptameri di ogni colore Sono stati sintetizzati molti aptameri di RNA che emettono per fluorescenza una grande varietà di colori. Questo è molto utile perché permette di marcare diversi tipi di RNA nella stessa cellula con colori diversi per poterli distinguere. Inoltre usando tecniche come FRET si possono monitorare anche le distanze tra i diversi fluorofori in una cellula vivente. Qui si possono vedere tre esempi di aptameri che emettono luce di fluorescenza di colore diverso: l'aptamero giallo Corn (file PDB 5bjp), l'aptamero arancione Mango-II (file PDB 6c63) e l'aptamero rosso DIR2s (file PDB 6db8) Esplorando la struttura Nell'immagine qui sotto si vede l'aptamero Spinacio (file PDB 4ts2) che circonda il suo fluoroforo (verde) formando una tasca rigida che aumenta la fluorescenza verde della molecola. La faccia inferiore del fluoroforo è appoggiata ad un quartetto di guanine G4 (mostrate in arancione). Nella struttura vi sono due quartetti di guanine arancioni consecutivi. La faccia superiore è a contatto con una tripletta di basi nucleotidiche (un'adenina e due uracili mostrati in magenta). Un'altra guanina (bianca) interagisce con il bordo del fluoroforo e lo mantiene in posizione nella tasca. 
 Il
quartetto di guanine G4 è una struttura planare complessa
formata da quattro guanine legate tra loro con otto legami idrogeno mostrati
con puntini verdi. Il
quartetto di guanine G4 è una struttura planare complessa
formata da quattro guanine legate tra loro con otto legami idrogeno mostrati
con puntini verdi. I legami idrogeno si formano tra la testa di una guanina e il fianco della guanina successiva (file PDB quartetto G4). Un quartetto simile si trova nell'enzima telomerasi (mdm 11-2018).  Anche
la tripletta di nucleotidi che si trova sopra il fluoroforo è
una struttura planare complessa. E' formata da due uracili e da una adenina
legati tra loro con quattro legami idrogeno. Quelli orizzontali tra uracile
e adenina sono i normali legami idrogeno testa a testa che si formano
tra le coppie di basi azotate in una doppia elica. Anche
la tripletta di nucleotidi che si trova sopra il fluoroforo è
una struttura planare complessa. E' formata da due uracili e da una adenina
legati tra loro con quattro legami idrogeno. Quelli orizzontali tra uracile
e adenina sono i normali legami idrogeno testa a testa che si formano
tra le coppie di basi azotate in una doppia elica. Quelli verticali sono realizzati in modo anomalo tra la testa dell'uracile in alto e il fianco dell'adenina (file PDB tripletta) Queste ultime due immagini sono state realizzate con MGL Tools (vedi Chimica al Computer). Spunti per ulteriori esplorazioni Per vedere come gli aptameri Spinacio sono impaccati nel cristallo, scegliete l'opzione 3D view: Structure nella pagina principale del sito RCSB e scegliete l'opzione Unit Cell o Supercell nel menù Assembly. La maggior parte degli aptameri hanno un quadruplesso G che si addossa al fluoroforo. Esplorando queste strutture provate ad osservare come è arrangiata la catena dell'RNA per mettere 4 guanine nel piano nella giusta posizione. Trachman, R. J., Truong, L., Ferré-D'Amaré, A. R. (2017) Structural principles of fluorescent RNA aptamers. Trends Pharmacol. Sci. 38: 928-939. 6db8: Shelke, S.A., Shao, Y., Laski, A., Koirala, D., Weissman, B.P., Fuller, J.R., Tan, X., Constantin, T.P., Waggoner, A.S., Bruchez, M.P., Armitage, B.A., Piccirilli, J.A. (2018) Structural basis for activation of fluorogenic dyes by an RNA aptamer lacking a G-quadruplex motif. Nat. Commun 9: 4542-4542. 6c63: Trachman 3rd., R.J., Abdolahzadeh, A., Andreoni, A., Cojocaru, R., Knutson, J.R., Ryckelynck, M., Unrau, P.J., Ferré-D'Amaré, A.R. (2018) Crystal structures of the Mango-II RNA aptamer reveal heterogeneous fluorophore binding and guide engineering of variants with improved selectivity and brightness. Biochemistry 57: 3544-3548. 5bjp: Warner, K.D., Sjekloca, L., Song, W., Filonov, G.S., Jaffrey, S.R., Ferré-D'Amaré, A.R. (2017) A homodimer interface without base pairs in an RNA mimic of red fluorescent protein. Nat. Chem. Biol. 13: 1195-1201. 4kzd: Huang, H., Suslov, N.B., Li, N.S., Shelke, S.A., Evans, M.E., Koldobskaya, Y., Rice, P.A., Piccirilli, J.A. (2014) A G-quadruplex-containing RNA activates fluorescence in a GFP-like fluorophore. Nat. Chem. Biol. 10: 686-691. 4ts2: Warner, K.D., Chen, M.C., Song, W., Strack, R.L., Thorn, A., Jaffrey, S.R., Ferré-D'Amaré, A.R. (2014) Structural basis for activity of highly efficient RNA mimics of green fluorescent protein. Nat. Struct. Mol. Biol. 21: 658-663.
|
|||||||
|
|
|||||||
