|
|
Ubiquitina |
|||||
|
Molecola del mese di dicembre 2004 L'ubiquitina serve per marcare le proteine obsolete per poi portarle alla distruzione nei proteasomi 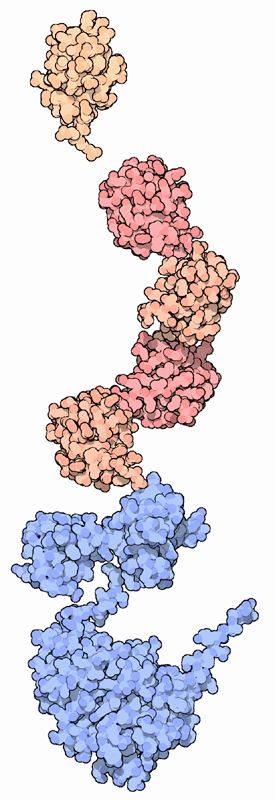 Introduzione IntroduzioneNiente dura per sempre. Molte proteine non durano più di qualche minuto. Le cellule sintetizzano continuamente nuove proteine, le usano per un solo compito e poi le eliminano. Per esempio, le proteine che vengono usate per segnalazione e controllo, come i regolatori della trascrizione e le cicline che controllano la divisione cellulare, hanno una vita molto breve, portano i loro messaggi e poi vengono eliminate. Anche gli enzimi specializzati nella sintesi di particolari molecole vengono costruiti solo quando sono necessari, e così permettono alla cellula di controllare quello che vuole sintetizzare momento per momento. Questa strategia di invecchiamento programmato può sembrare uno spreco, ma permette alla cellula di rispondere rapidamente ad ogni nuova necessità. Eliminare il vecchio Naturalmente le cellule hanno bisogno di controllare la distruzione delle proprie proteine, per eliminare solo quelle che non sono più necessarie. La piccola proteina ubiquitina ha un ruolo centrale in questo meccanismo. L'ubiquitina si lega alle proteine inutili, segnalando alla cellula che sono pronte per essere degradate. Come si vede nella figura qui a lato, una sequenza di molecole di ubiquitina rosa e beige (file PDB 1ubq) viene legata ad una proteina obsoleta blu (file PDB 2src). L'ubiquitina viene poi riconosciuta dall'apparato di distruzione della cellula. Ubiquitina ubiquitaria Come dice il suo nome, l'ubiquitina si trova in tutte le cellule eucariote e quindi anche in ogni cellula del nostro corpo. Il Premio Nobel per la chimica di quest'anno è stato assegnato ai tre ricercatori che hanno scoperto la sua funzione essenziale nel 1980. Negli anni seguenti, è divenuto chiaro che l'ubiquitina oltre al suo ruolo di marcatore di proteine ha anche altri scopi, come dirigere il trasporto di proteine dentro e fuori dalla cellula. Connettendo insieme più ubiquitine in catene corte o lunghe, o usando connessioni diverse tra le molecole, possono essere codificati segnali molto diversi. A causa dei compiti importanti che svolge, l'ubiquitina è cambiata molto poco nel corso dell'evoluzione, così si possono trovare ubiquitine quasi identiche nelle cellule di lievito, in quelle vegetali e anche nelle nostre.  Ubiquitinazione Ubiquitinazione
Il segreto di questo processo sta nell'assicurarsi che l'ubiquitina si leghi solo alle giuste proteine. Molti enzimi specializzati passano in rassegna le proteine nella cellula e scelgono quelle che devono essere eliminate. Ci sono tre tipi di questi enzimi, chiamati E1, E2, ed E3. L'enzima E1, mostrato qui a fianco in alto (file PDB 1r4n) è l'enzima che attiva l'ubiquitina e avvia il processo. Utilizzando l'energia di un ATP (rosso), si lega sulla coda dell'ubiquitina (beige) con uno dei propri amminoacidi, una cisteina (verde; in questa struttura, la cisteina è stata sostituita con un'alanina). Poi l'enzima E2 prende da E1 l'ubiquitina attivata e la unisce alle proteine (file PDB 1fxt). Una serie di enzimi E3 diversi lavorano insieme con gli enzimi E2 per riconoscere la proteine inutili e per legarle all'ubiquitina. L'enzima E3 mostrato qui (file PDB 1ldk e 1fqv) ha la forma di una tenaglia. La proteina bersaglio viene legata nell'apertura indicata da un asterisco. Il lato sinistro dell'enzima riconosce la proteina ed il lato destro posiziona E2 per permettere il trasferimento dell'ubiquitina.  Distruzione
totale Distruzione
totale Le proteine che sono diventate inutili vengono dapprima marcate con almeno quattro molecole di ubiquitina e poi vengono distrutte dai proteasomi. I proteasomi, anche chiamati AAA+ Proteasi (mdm 8-2006), sono insaziabili frantumatori di proteine, ma il loro apparato distruttivo è nascosto all'interno di una struttura cilindrica in modo che non possa attaccare le normali proteine nella cellula. Il proteasoma mostrato qui a destra (file PDB 1fnt) ha la forma di un cilindro, coi suoi siti attivi che tagliano le proteine situati all'interno del tubo. I due tappi agli estremi controllano l'entrata nella camera di distruzione, dove le proteine vengono tagliate in pezzi lunghi da 3 a 23 amminoacidi. Esplorando la Struttura La figura qui sotto (file PDB 1f9j) mostra come le molecole di ubiquitina si legano insieme per formare sequenze di ubiquitine. Anche se il cristallo che ha generato questa struttura è stato prodotto con tetraubiquitina (quattro proteine concatenate), le molecole di ubiquitina sono legate a due a due con un legame covalente. I legami, mostrati con sfere colorate, uniscono la parte terminale di una catena (gialla) con un punto al centro dell'altra catena (rossa). .. . . . . ..  Nell'immagine ravvicinata qui sotto, si può vedere che i legami tra le catene sono insoliti. L'ultimo amminoacido della catena gialla, glicina (Gly), con il suo carbossile alfa terminale (C bianco, O rosso) è legato al gruppo amminico (N blu) della catena laterale di una lisina (Lys) che si trova al centro della catena rossa. Si tratta di un legame ammidico anomalo, infatti i normali legami ammidici delle proteine uniscono tra loro il carbossile alfa di un amminoacido con il gruppo amminico alfa dell'amminoacido successivo senza mai coinvolgere gruppi in catena laterale. In questo modo le proteine formano sempre catene lineari, mentre qui si è formata una ramificazione. .. . . . . . 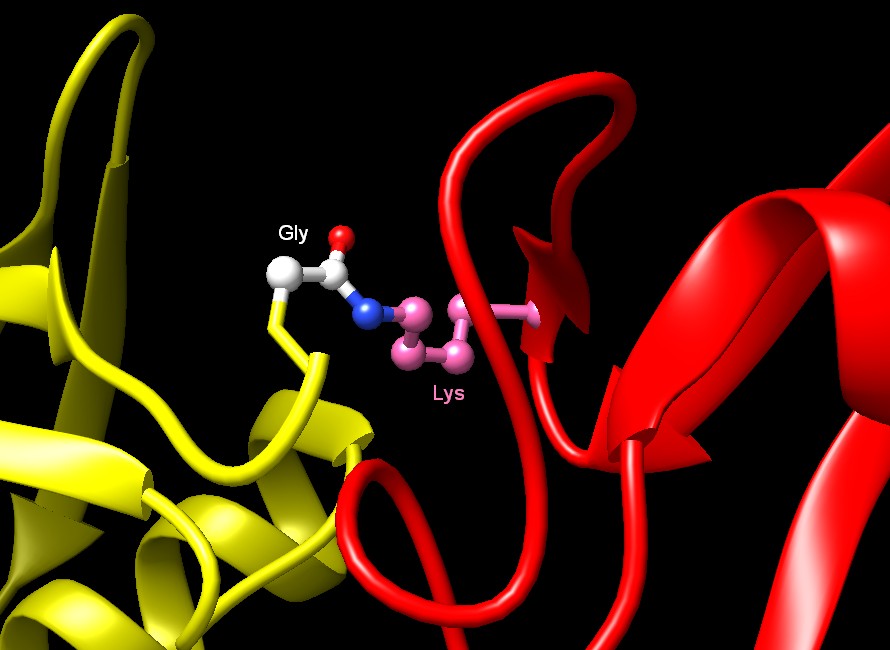 A. P. VanDemark and C. P. Hill (2002) Structural Basis of Ubiquitylation. Current Opinion in Structural Biology 12, 822-830. M. H. Glickman and A. Ciechanover (2002) The Ubiquitin-Proteasome Proteolytic Pathway: Destruction for the Sake of Construction. Physiological Reviews 82, 373-428. C. M. Pickart (2000) Ubiquitin in Chains. Trends in Biochemical Sciences 25, 544-548.
|
||||||
|
|
||||||
