|
|
SARS-CoV2 proteina spike |
|||||
|
Molecola del mese di giugno 2020 La proteina spike del Coronavirus si lega alla superficie delle cellule ed è un bersaglio per lo sviluppo del vaccino  Introduzione IntroduzioneLa comunità scientifica si è subito mobilitata per combattere il virus della pandemia SARS-CoV2 sfruttando l'esperienza accumulata in anni di lavoro contro il precedente virus SARS-CoV. La proteina spike del virus si sta rivelando fondamentale in questa lotta perché è il bersaglio principale degli anticorpi che si sviluppano nelle persone guarite che hanno sviluppato immunità contro il virus. La superficie dei coronavirus è coperta di queste proteine spike (punte) che danno loro la caratteristica forma a corona nelle immagini al microscopio elettronico. Le proteine spike iniziano il processo di infezione legandosi a recettori di membrana e consentendo al virus di fondersi con la membrana cellulare e di iniettare all'interno della cellula l'RNA genomico virale. Molti altri virus usano proteine simili alle spike per infettare le cellule: il virus dell'influenza usa l'emoagglutinina (mdm 4-2006), mentre i virus dell'HIV-1 e di ebola usano speciali glicoproteine di superficie. Tagliare nelle giuste dimensioni La proteina spike è formata da tre catene identiche che formano un complesso con un piccolo dominio interno al virus, un segmento che attraversa la membrana ed un dominio più grande che si estende fuori dal virus. Inoltre, le proteina spike è una glicoproteina: il suo dominio esterno è coperto di catene di zuccheri che servono a mascherarla contro il sistema immunitario. Qui sopra sono mostrate le strutture del dominio esterno delle proteine spike dei due virus SARS-CoV (nella forma estesa) e SARS-CoV2 (nella forma chiusa) (file PDB 6crz e 6vxx) e, come potete vedere, sono molto simili. Queste proteine vengono sintetizzate come un un'unica catena che poi viene tagliata dalle proteasi cellulari dell'ospite in due frammenti. Il segmento S1, magenta, si lega ai recettori di membrana della cellula, mentre il segmento S2, rosso, guida la fusione del virus con la cellula. Entrambe le strutture contengono solo una parte delle molte catene di zuccheri (di colore più chiaro) che ricoprono la superficie della proteina spike perché gli zuccheri sono molto flessibili e quindi sono difficili da osservare. Inportanza della flessibilità Recenti studi hanno rivelato che le proteine spike di SARS-CoV e di SARS-CoV2 sono molto flessibili. In queste strutture i domini che si legano ai recettori assumono diverse conformazioni. Spesso vi è un solo dominio esteso in fuori, ma talvolta entrambi sono ripiegati in basso oppure sono entrambi estesi. La conformazione estesa è indispensabile per legarsi ai recettori, quindi la flessibilità è un grande vantaggio per il virus. I ricercatori credono che la particolare virulenza delle varianti recenti di SARS sia dovuta alla grande flessibilità dei domini che si legano ai recettori, mentre i coronavirus che causano il normale raffreddore sono meno pericolosi perché hanno domini meno flessibili. 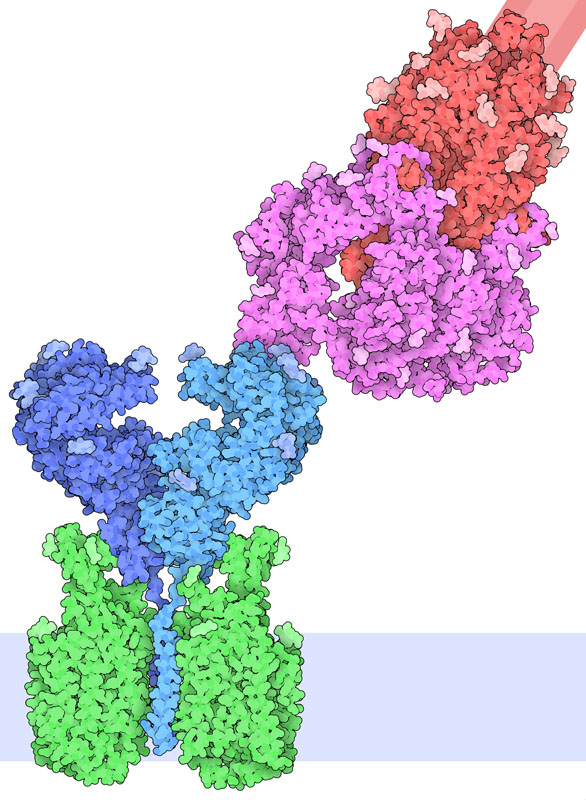 Legare
il recettore Legare
il recettoreLa proteina spike si lega ad ACE2 (angiotensin-converting enzyme-2) sulla superficie della cellula. ACE2 è un enzima che attiva la angiotensina, un ormone che controlla la pressione del sangue. ACE2 si trova nelle cellule del fegato, del cuore, dei reni e dell'intestino e infatti sono queste le cellule bersaglio dell'infezione virale. Nella struttura mostrata qui a fianco, si vede il complesso tra ACE2 (blu chiaro e scuro) e la proteina spike (magenta e rossa) che si lega usando il dominio magenta proteso in fuori. ACE2 è a sua volta legata al trasportatore di amminoacidi B0AT1 (verde), una proteina di membrana. Questa immagine è stata ottenuta combinando la struttura del complesso ACE2- B0AT1 (file PDB 6m17) con quella della proteina spike nella forma aperta (file PDB 6vsb). La membrana cellulare, azzurra in basso, è rappresentata in modo schematico  Esplorando
la struttura Esplorando
la strutturaIl nostro sistema immunitario attacca il coronavirus quando questo ci infetta. La proteina spike è il bersaglio principale di questo attacco perché è esposta sulla superficie del virus. In queste tre strutture (file PDB 3bgf, 2ghw e 6w41) si vede che gli anticorpi (verdi) possono riconoscere le proteine spike in modi diversi. Due di questi anticorpi bloccano il dominio che lega il recettore (mostrato in colori più scuri), ma il terzo anticorpo si lega ad un sito alla base del dominio che normalmente è nascosto fino a quando l'anticorpo non lo lega. Spunti per ulteriori esplorazioni Per avere altre informazioni su SARS-CoV2 and COVID-19, visitate la pagina risorse nel sito RCSB PDB 6vxx: Walls, A.C., Park, Y.J., Tortorici, M.A., Wall, A., McGuire, A.T., Veesler, D. (2020) Structure, Function, and Antigenicity of the SARS-CoV-2 Spike Glycoprotein. Cell 181, 281-292 6w41: Yuan, M., Wu, N.C., Zhu, X., Lee, C.D., So, R.T.Y., Lv, H., Mok, C.K.P., Wilson, I.A. (2020) A highly conserved cryptic epitope in the receptor-binding domains of SARS-CoV-2 and SARS-CoV. Science DOI: 10.1126/science.abb7269 6vsb: Wrapp, D., Wang, N., Corbett, K.S., Goldsmith, J.A., Hsieh, C.L., Abiona, O., Graham, B.S., McLellan, J.S. (2020) Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation. Science 367: 1260-1263 6m17: Yan, R., Zhang, Y., Li, Y., Xia, L., Guo, Y., Zhou, Q. (2020) Structural basis for the recognition of SARS-CoV-2 by full-length human ACE2. Science 367: 1444-1448 6crz: Kirchdoerfer, R.N., Wang, N., Pallesen, J., Wrapp, D., Turner, H.L., Cottrell, C.A., Corbett, K.S., Graham, B.S., McLellan, J.S., Ward, A.B. (2018) Stabilized coronavirus spikes are resistant to conformational changes induced by receptor recognition or proteolysis. Sci Rep 8: 15701-15701 3bgf: Pak, J.E., Sharon, C., Satkunarajah, M., Auperin, T.C., Cameron, C.M., Kelvin, D.J., Seetharaman, J., Cochrane, A., Plummer, F.A., Berry, J.D., Rini, J.M. (2009) Structural insights into immune recognition of the severe acute respiratory syndrome coronavirus S protein receptor binding domain. J Mol Biol 388: 815-823 2ghw: Hwang, W.C., Lin, Y., Santelli, E., Sui, J., Jaroszewski, L., Stec, B., Farzan, M., Marasco, W.A., Liddington, R.C. (2006) Structural basis of neutralization by a human anti-severe acute respiratory syndrome spike protein antibody, 80R. J Biol Chem 281: 34610-34616
|
||||||
|
|
||||||
