|
|
Ribonucleasi P |
|||||
|
Molecola del mese di agosto 2021 La ribonucleasi P è un ribozima che taglia il pre-tRNA per formare tRNA maturo 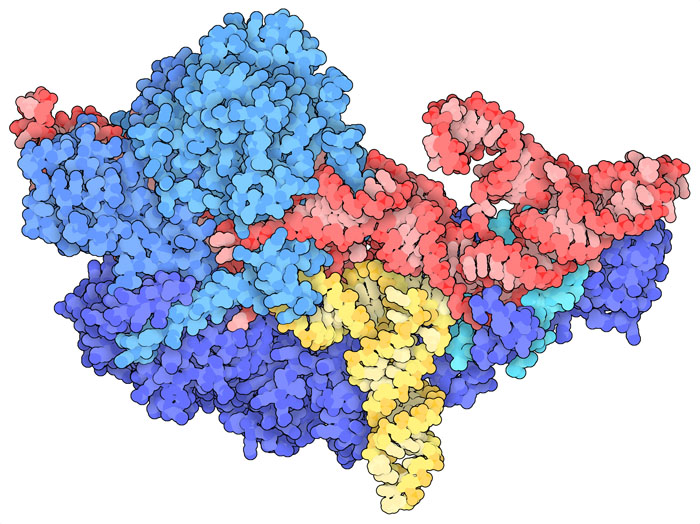 Ribozimi RibozimiLa maggior parte delle reazioni chimiche che avvengono nelle cellule sono catalizzate da enzimi proteici. In alcuni casi particolari, però, gli enzimi sono costituiti da RNA e vengono chiamati ribozimi. Per esempio, l'auto-splicing RNA (mdm 5-2005) si ritaglia da solo a partire da un filamento più lungo di RNA appena trascritto senza l'aiuto di enzimi proteici. Altri esempi di ribozimi sono quelli che hanno un ruolo chiave nella biosintesi delle proteine: la ribonucleasi P (RNasi P), l'RNasi MRP e i ribosomi (mdm 1-2010). La RNasi P taglia il pre-tRNA producendo tRNA (RNA transfer) (mdm 3-2001) con la corretta terminazione 5'. La RNAasi MRP fa dei tagli all'RNA dei ribosomi appena sintetizzati. I ribosomi sintetizzano le proteine con una reazione catalizzata dall'RNA ribosomiale. Tagliare il tRNA Il transfer RNA partecipa alla sintesi delle proteine portando il giusto amminoacido in corrispondenza di ogni codone di tre nucleotidi sull'mRNA. Dato che questo processo è fondamentale per la sopravvivenza della cellula, le molecole di tRNA devono essere molto affidabili per questo i pre-tRNA formati dalla trascrizione sono sottoposti ad alcune modifiche strutturali. La endonucleasi RNasi P taglia un piccolo segmento dall'estremità 5' del pre-RNA producendo una molecola di tRNA maturo, pronto per svolgere il suo lavoro. La RNasi P umana nucleare è formata da 11 proteine e da un grande filamento di RNA che svolge l'azione catalitica. Le 11 proteine hanno la funzione di stabilizzare e orientare l'RNA in modo che possa riconoscere il pre-tRNA e svolgere l'azione catalitica. RNasi P diverse Mentre i batteri e gli archea hanno un solo tipo di RNasi P, alcuni eucarioti ne hanno di due tipi. Un ribozima RNasi P formato da proteine e da un filamento di RNA catalitico, ed una forma del tutto priva di RNA, chiamata PRORP (protein only RNasi P). Anche se le due endonucleasi svolgono la stessa funzione, sono diverse sia per struttura che per ubicazione nella cellula. PRORP opera nei mitocondri, dove taglia il pre-tRNA coinvolto nell'espressione dei geni che presiedono alla produzione di energia e che si trovano nel DNA mitocondriale. La RNasi P basata sull'RNA, invece, modifica il pre-tRNA nel nucleo della cellula. Le proteine della PRORP e della RNasi P basata sull'RNA svolgono funzioni simili, ma hanno strutture diverse. 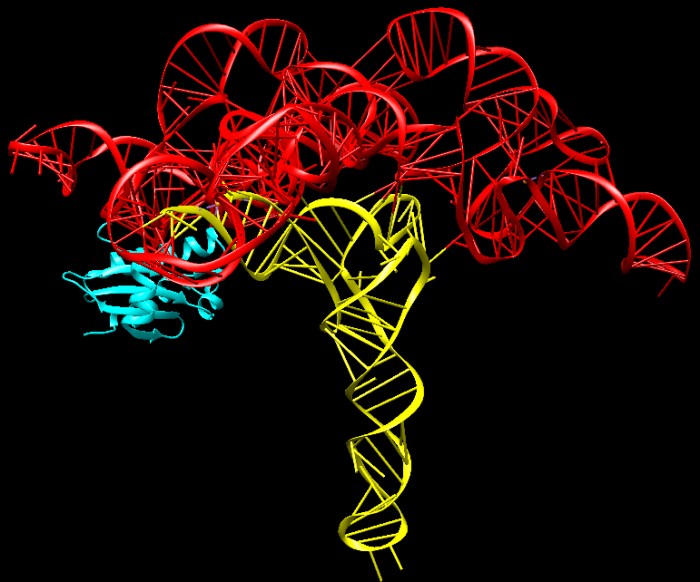 Fossile
molecolare Fossile
molecolareL'RNasi P è uno dei due ribozimi che sono presenti in quasi tutti gli organismi, l'altro è il ribosoma. La cosa notevole è che il filamento di RNA della RNasi P (mostrato in rosso nelle figure qui a lato) è molto simile nei batteri, negli archea e negli eucarioti. Questo significa che la RNasi P era presente prima che l'evoluzione diversificasse i tre regni degli organismi viventi e quindi si è sviluppata nelle primissime forme di vita apparse sul pianeta terra, basate sull'RNA. Le differenze maggiori sono nelle proteine che completano l'enzima: gli organismi più complessi contengono un numero maggiore di subunità proteiche. Nella figura qui a lato si vede la RNasi P batterica (file PDB 3q1q) che contiene una piccola subunità proteica (ciano) che costituisce solo il 10% della massa totale. La catena di RNA è mostrata in rosso, mentre quella del tRNA è gialla. 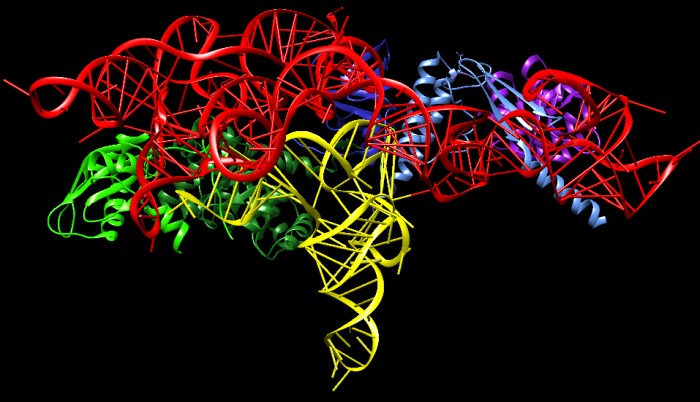 Qui a fianco è mostrata la RNasi P di un archea, il metanococco,
(file PDB 6k0b) che contiene cinque subunità
proteiche evidenziate con colori dal verde al viola.
Qui a fianco è mostrata la RNasi P di un archea, il metanococco,
(file PDB 6k0b) che contiene cinque subunità
proteiche evidenziate con colori dal verde al viola. Infine
qui a lato è mostrata la RNasi P umana nucleare (file PDB
6ahu) che contiene undici subunità
proteiche che circondano l'RNA catalitico (rosso) e lo aiutano a svolgere
al meglio la sua azione sul tRNA (giallo). Infine
qui a lato è mostrata la RNasi P umana nucleare (file PDB
6ahu) che contiene undici subunità
proteiche che circondano l'RNA catalitico (rosso) e lo aiutano a svolgere
al meglio la sua azione sul tRNA (giallo).Esplorando la struttura Ribonucleasi P in azione L'uracile 80 (U80) è un nucleotide conservato in tutte le RNasi e si trova nel sito catalitico del ribozima. Lo studio dei nucleotidi del sito attivo della RNasi P umana ha rivelato che si verifica un cambiamento di conformazione nell'U80 quando si lega il tRNA. 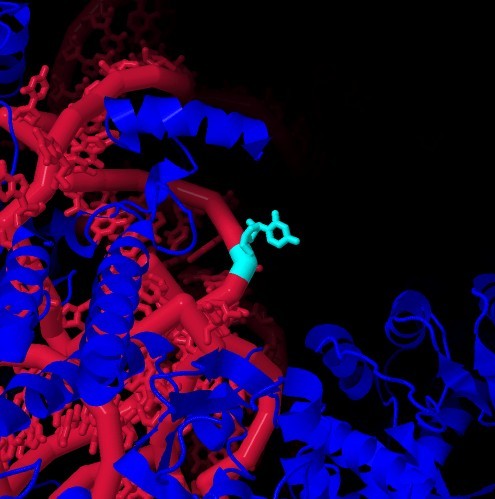
 Confrontando le due strutture dell'enzima con e senza tRNA, qui sopra a sinistra si vede che l'U80 (ciano) sporge della catena in assenza di tRNA (file PDB 6ahu) creando un ingombro sterico che impedisce la catalisi. Qui sopra a destra si vede che, quando l'enzima si lega al tRNA, l'U80 deve piegarsi all'interno e innesca la catalisi (file PDB 6ahr). Spunti per ulteriori esplorazioni Negli archivi PDB sono disponibili molte strutture di RNase MRP come, per esempio, 6w6v. Gli enzimi PRORP sono RNasi prive della subunità catalitica di RNA. Esaminate la struttura tridimensionale di un enzima PRORP al file PDB 4g24. 6k0b: Wan, F., Wang, Q., Tan, J., Tan, M., Chen, J., Shi, S., Lan, P., Wu, J., Lei, M. (2019) Cryo-electron microscopy structure of an archaeal ribonuclease P holoenzyme. Nature Communications 10: 2617-2617 6ahr, 6ahu : Wu, J., Niu, S., Tan, M., Huang, C., Li, M., Song, Y., Wang, Q., Chen, J., Shi, S., Lan, P., Lei, M. (2018) Cryo-EM Structure of the Human Ribonuclease P Holoenzyme. Cell 175, 1393-1404. Klemm, B.P., Wu, N., Chen, Y., Liu, X., Kaitany, K.J., Howard, M.J., Fierke, C.A. (2016) The diversity of ribonuclease P: Protein and RNA catalysts with analogous biological functions. Biomolecules 6: 27. 3q1q: Reiter, N., Osterman, A., Torres-Larios A., Swinger K.K., Pan T., Mondragon, A. (2010) Structure of a bacterial ribonuclease P holoenzyme in complex with tRNA. Nature 468, 784-789. Sun F.J., Caetano-Anollés, G. (2010) The ancient history of the structure of ribonuclease P and the early origins of Archaea. BMC Bioinformatics 11: 153. Holzmann, J., Frank, P., Loffler, E., Bennett, K.L., Gerner, C., Rossmanith, W. (2008) RNase P without RNA: Identification and functional reconstitution of the human mitochondrial tRNA processing enzyme. Cell 135: 462-474. Walker, S.C., Engelke, D.R. (2008) A protein-only RNase P in human mitochondria. Cell 135:412-414. Cech, T.R. (2002) Ribozymes, the first 20 years. Biochem Soc Trans 30: 1162-1166.
|
||||||
|
|
||||||
