|
Molecola del mese
di agosto 2023
Le cellule che si dividono usano gli enzimi ATM e ATR chinasi per riparare
i danni al DNA
 Introduzione Introduzione
Quando le cellule si dividono, devono controllare che il DNA venga copiato
in modo completo ed accurato.
Alcuni fattori ambientali come le radiazioni UV e le sostanze tossiche
possono danneggiare il DNA, ma anche nei normali processi di duplicazione
del DNA possono avvenire degli errori. Nel DNA di ogni cellula si possono
verificare decine di migliaia di errori al giorno. Se le cellule non
riescono a trovare e a correggere questi errori, si può sviluppare
il cancro. Le nostre cellule dispongono di tecniche molto sofisticate
per controllare il DNA e riconoscere quando è danneggiato e così
sospendono la divisione cellulare fino a quando il DNA non è
riparato.
Le proteina chinasi ATM (Ataxia-telangiectasia mutated) e ATR (Ataxia
telangiectasia and Rad3-related protein) sono dei regolatori essenziali
dei processi si controllo e riparazione del DNA. Queste due proteine
sono state identificate durante lo studio della Ataxia telangiectasia,
una malattia neurodegenerativa che comporta anche disfunzione del sistema
immunitario, aumento di sensibilità alle radiazioni e predisposizione
al cancro. Quando individuano un danno al DNA, le chinasi ATM e ATR
lavorano insieme per fermare temporaneamente la divisione cellulare
e inducono altre proteine a riparare il danno.
Chiedere aiuto
Le chinasi ATM e ATR segnalano alla cellula vari tipi di danni al DNA.
ATM segnala le rotture della doppia elica che sono un grave pericolo
per la sopravvivenza della cellula, mentre ATR identifica le catene
di DNA a singolo filamento nelle quali anche una sola rottura non riparata
può essere sufficiente ad uccidere la cellula.
Durante la duplicazione del DNA, la DNA elicasi separa le due catene
del DNA formando regioni a singolo filamento che servono da stampo per
la DNA polimerasi. Se però l'attività della DNA polimerasi
si arresta, la DNA elicasi può continuare a srotolare la doppia
elica formando tratti di DNA a singolo filamento pericolosamente lunghi.
Singoli filamenti di DNA si formano anche nelle catene rotte di DNA
durante il processo di riparazione quando i terminali vengono tagliati
prima di essere riconnessi.
Quando ATM e ATR individuano un danno nel DNA, fosforilano e quindi
attivano centinaia di proteine coinvolte nel processo di duplicazione
e riparazione del DNA. Tra queste vi è il soppressore
di tumore p53 (mdm 7-2002) e la proteina RAD51 coinvolta nella
ricombinazione omologa, un processo di riparazione ad alta fedeltà
che usa come stampo il cromosoma omologo per riparare il tratto corrispondente
del cromosoma danneggiato.
Strutture complesse
Gli scienziati hanno cominciato a capire il meccanismo molecolare di
segnalazione di ATM e ATR grazie alle strutture ottenute con la microscopia
crioelettronica.
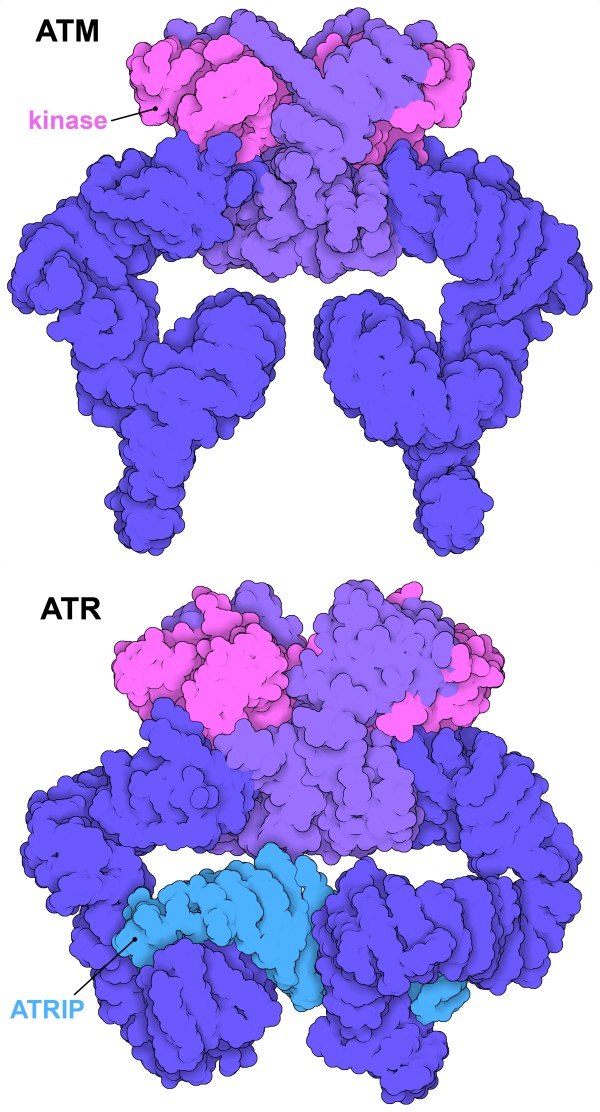
Qui sopra sono mostrate le strutture 3D della ATM (file PDB 5np0)
e della ATR (file PDB 5yz0). ATM e ATR
appartengono alla stessa famiglia di proteine e formano grandi complessi
a forma di farfalla con molte parti funzionali.
Il dominio di chinasi (magenta) è solo una piccola parte del
complesso e realizza la reazione di fosforilazione.
Il resto della proteina (in due gradazioni di viola) ha molti domini
che mediano l'interazione con specifici sensori del DNA danneggiato
e con le proteine attivate dalla fosforilazione. In azzurro è
mostrata ATRIP, cioè ATR interactive protein, una proteina che
aiuta ATR ad interagire con le proteine che legano il DNA a singolo
filamento.
 Riparazione
del DNA Riparazione
del DNA
Le chinasi ATM e ATR sono i messaggeri chiave nella risposta ai danni
al DNA, ma si affidano anche ad altre proteine per individuare il DNA
danneggiato. Due di queste sono mostrate qui a fianco.
La prima, mostrata in basso, è il complesso di Mre11 e Rad50
(file PDB 5dny e 5gox)
che è legato ad una catena spezzata di DNA a doppio filamento
(giallo).
La seconda, sulla destra, è RPA o proteina di replicazione A
(file PDB 4gnx) che è legata ad
una catena di DNA a singolo filamento (giallo).
Queste proteine interagiscono rispettivamente con ATM e ATR attivando
i loro domini di chinasi e così le inducono ad attivare, a loro
volta, una cascata di altre proteine coinvolte nel processo di riparazione
del DNA.
 Esplorando
la struttura Esplorando
la struttura
Dato che hanno un ruolo centrale nella riparazione del DNA, le chinasi
ATM e ATR sono fortemente coinvolte nello sviluppo del cancro. Molto
spesso le cellule cancerose hanno delle varianti mutate di ATM e ATR
che non sono in grado di fermare la divisione cellulare quando il genoma
è danneggiato.
I ricercatori stanno cercando di sfruttare questo fatto progettando
farmaci che bloccano ATM da usare nelle terapie antitumorali.
Se questi farmaci vengono iniettati nei tumori, li rendono più
sensibili alla radioterapia perchè diventano incapaci di riparare
i danni al DNA provocati dalle radiazioni.
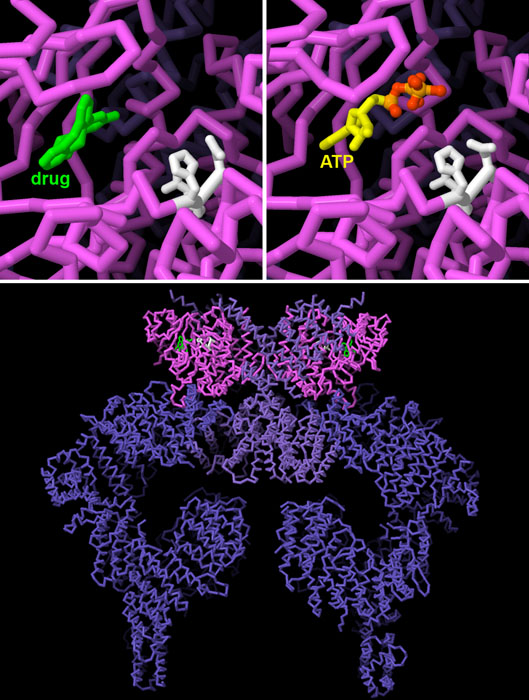
Nell'immagine qui a fianco sulla sinistra (file PDB 7ni5)
si può vedere la proteina ATM legata ad uno di questi farmaci
sperimentali. Il farmaco (verde) si lega nel sito attivo della chinasi
nello stesso punto dove si lega ATP (giallo, arancione e rosso), come
è mostrato nella figura sulla destra (file PDB 7ni6).
Notate che ai margini del sito attivo vi sono i tre amminoacidi (bianchi)
che catalizzano la reazione di fosforilazione.
In presenza del farmaco inibitore, l'enzima ATM non si può legare
ad ATP e quindi non può svolgere la sua azione di chinasi.
Spunti per ulteriori esplorazioni
Agli enzimi chinasi ATM e ATR sono stati assegnati nomi diversi in organismi
diversi. Esaminate, per esempio, le proteine di lievito Tel1 (file PDB
6s8f) o Mec1 (file PDB 6z3a)
che sono rispettivamente ATM e ATR.
 Bibliografia Bibliografia
Phan, L. M., Rezaeian, A. H. (2021) ATM: Main features, signaling
pathways, and its diverse toles in DNA damage response, tumor suppression,
and cancer development. Genes 12, 845.
7ni5, 7ni6: Stakyte, K., Rotheneder, M., Lammens, K., Bartho,
J. D., Gradler, U., Fuchs, T., Pehl, U., Alt, A., van de Logt, E., Hopfner,
K. P. (2021) Molecular basis of human ATM kinase inhibition. Nat Struct
Mol Biol 28, 789-798.
6z3a: Tannous, E.A., Yates, L.A., Zhang, X., Burgers, P. M. (2021)
Mechanism of auto-inhibition and activation of Mec1ATR checkpoint kinase.
Nat Struct Mol Biol 28, 50-61.
Williams, R. M., Yates, L. A., Zhang, X. (2020) Structures and
regulations of ATM and ATR, master kinases in genome integrity. Curr
Opin Struct Bio 61, 98-105.
Sun, Y., McCorvie, T. J., Yates, L. A., Zhang, X. (2019) Structural
basis of homologous recombination. Cell Molec Life Sci 77, 3-18.
5np0: Baretic, D., Pollard, H.K., Fisher, D.I., Johnson, C.M.,
Santhanam, B., Truman, C.M., Kouba, T., Fersht, A.R., Phillips, C.,
Williams, R.L. (2017) Structures of closed and open conformations of
dimeric human ATM. Sci Adv 3: e1700933-e1700933.
5gox: Park, Y.B., Hohl, M., Padjasek, M., Jeong, E., Jin, K.S.,
Krezel, A., Petrini, J.H.J., Cho, Y. (2017) Eukaryotic Rad50 functions
as a rod-shaped dimer. Nat Struct Mol Biol 24: 248-257.
Rao, Q., Liu, M., Tian, Y., Wu, Z., Hao, Y., Song, L., Qin, Z.,
Ding, C., Wang, H.-W., Wang, J., Xu, Y. (2017) Cryo-EM structure of
human ATR-ATRIP complex. Cell Research 28, 143-156.
5dny: Liu, Y., Sung, S., Kim, Y., Li, F., Gwon, G., Jo, A., Kim,
A.K., Kim, T., Song, O.K., Lee, S.E., Cho, Y. (2016) ATP-dependent DNA
binding, unwinding, and resection by the Mre11/Rad50 complex. EMBO J
35: 743-758.
Weber, A. M., Ryan, A. J. (2015) ATM and ATR as therapeutic targets
in cancer. Pharmacology & therapeutics, 149, 124-138.
Maréchal, A., Zou, L. (2013) DNA damage sensing by the
ATM and ATR kinases. Cold Spring Harbor Persp Biol 5, a012716.
|
|
|