|
|
Proteasi-elicasi |
|||||
|
Molecola del mese di dicembre 2020 Le strutture delle proteine dell'epatite C virus hanno portato alla scoperta di antivirali ad azione diretta 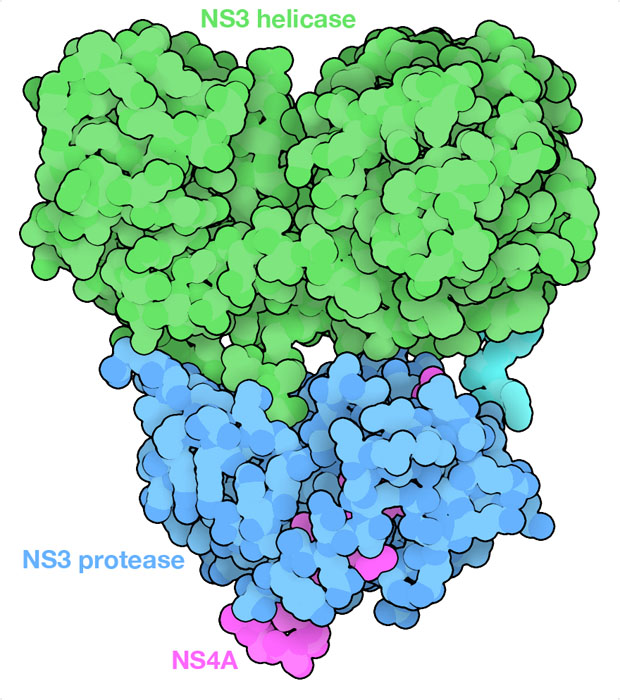 Introduzione IntroduzioneSe si conoscono i nemici è più facile sconfiggerli. La scoperta del virus dell'epatite C nel 1989 è un esempio perfetto di come la conoscenza scientifica si possa tradurre in un successo clinico. Prima di allora le trasfusioni di sangue comportavano un certo rischio di contrarre l'epatite, una malattia cronica che provoca gravi danni al fegato e persino tumori. Con la scoperta dell'agente responsabile della malattia, si sono messe a punto tecniche per accertarsi che il sangue non contenga il virus. Inoltre, sono state scoperte nuove terapie per combattere il virus nei pazienti infetti, così ora, finalmente, questa infezione cronica può essere curata. Per questo notevole successo della ricerca è stato attribuito il premio Nobel 2020 per la medicina e la fisiologia a H. J. Alter, M. Houghton, e C. M. Rice. Colpire l'HCV Come molti virus a RNA, l'epatite C virus (HCV) inganna i ribosomi della cellula ospite e li induce a sintetizzare una lunga proteina virale che deve poi essere tagliata nei frammenti funzionali da una proteasi virale, come accade con l'HIV proteasi (mdm 6-2000) e con la coronavirus proteasi (mdm 2-2020). Dato che queste proteasi sono fondamentali per il ciclo vitale del virus, sono dei bersagli ideali per i farmaci antivirali. Come si vede nell'immagine qui sotto, i ricercatori hanno sviluppato inibitori sempre più specifici per l'HCV proteasi e così oggi vi sono potenti antivirali ad azione diretta (DAA) che bloccano la crescita del virus e permettono di curare i pazienti infetti. Questa cura si è dimostrata preziosa per la salute globale perché si valuta che oltre 200 milioni di persone nel mondo siano infettate dall'HCV e questa malattia è la causa principale di trapianto di fegato. Due in uno La HCV proteasi è una proteasi alla serina simile alla chimotripsina (mdm 10-2003) e costituisce una parte di una proteina più lunga che contiene anche l'enzima elicasi (mdm 12-2013), indispensabile per la duplicazione dell'RNA virale genomico. Questa proteina composita è chiamata NS3 (proteina non strutturale 3) ed è mostrata qui sopra (file PDB 1cu1) con il dominio proteasi in blu ed elicasi in verde. Nella figura si vede anche un'altra piccola proteina virale, NS4A magenta, legata alla proteasi, che è necessaria per il corretto funzionamento della proteasi e serve anche a tenere il complesso legato alla membrana cellulare. 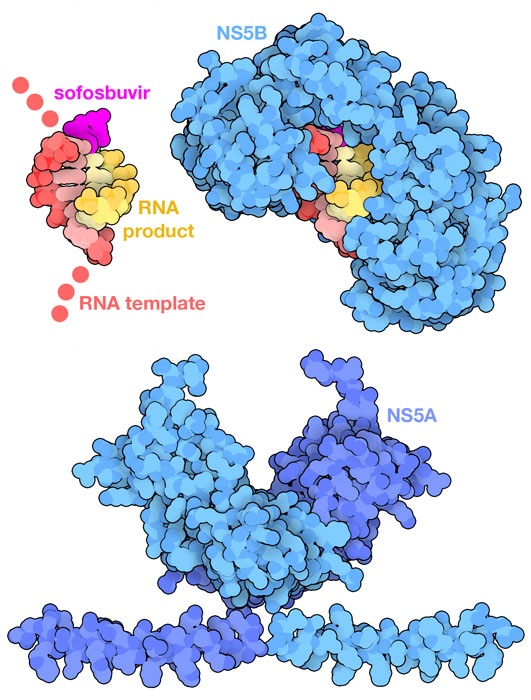 Bersagli
dei farmaci DAA Bersagli
dei farmaci DAAI ricercatori hanno cercato di colpire anche altre proteine dell'HCV con dei farmaci ad azione diretta. Come SARS-CoV-2, anche HCV ha un genoma di RNA che viene replicato da una RNA polimerasi RNA dipendente (mdm 9-2020) che è chiamata NS5B, mostrata qui a fianco (file PDB 4wtg). Questa struttura include anche il farmaco antivirale sofosbuvir (magenta) legato alla polimerasi e ad un breve tratto di RNA duplex. Sofosbuvir è un analogo modificato di un nucleotide di uracile che, quando viene incorporato nella catena di RNA ne provoca la terminazione. La sua struttura è mostrata qui sotto, sul carbonio 2 del ribosio, al posto di un OH, è legato un atomo di fluoro (verde) e inoltre vi è un atomo di carbonio extra (sferetta beige). Altri farmaci antivirali efficaci sono stati sviluppati per NS5A, una proteina dimera (qui a destra, blu e azzurra) che si lega alla membrana cellulare e ha un ruolo fondamentale per la replicazione dell'RNA e per l'assemblaggio del virus (File PDB 1zh1 per la parte centrale e 1r7g per la piccola catena che si lega alla membrana). Questi farmaci agiscono impedendo la dimerizzazione della proteina, bloccandone così la funzione .  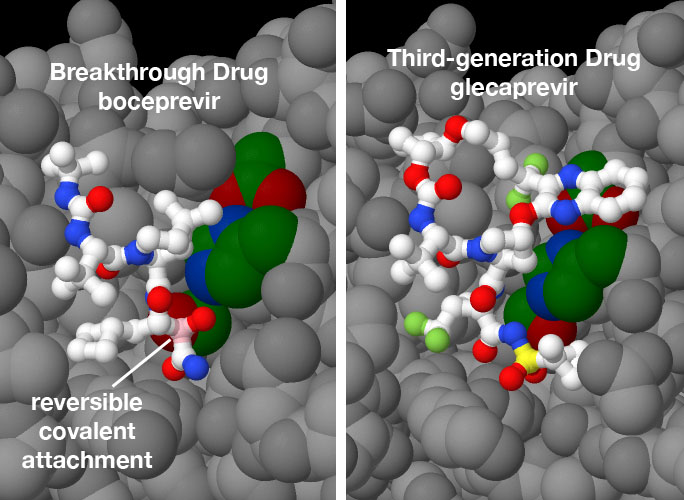 Esplorando
la struttura Esplorando
la strutturaIl sito attivo dell'HCV proteasi è un bersaglio conveniente perché non si trova in una tasca profonda come accade nella maggior parte degli enzimi. I primi farmaci che si sono rivelati efficaci nel bloccare il sito attivo della HCV proteasi sono stati boceprevir e telaprevir (file PDB 2oc8 e 3sv6) che formano legami covalenti reversibili con un amminoacido di serina nel sito attivo e così restano legati alla superficie dell'enzima. Sfortunatamente, però, questi primi farmaci provocano anche effetti collaterali indesiderati e inoltre la loro efficacia non è duratura perchè il virus sviluppa in breve tempo resistenza. La lotta contro HCV si è rivelata una sfida difficile a causa delle continue mutazioni del virus di cui si conoscono ormai almeno 86 varianti. La ricerca è continuata fino a produrre gli attuali farmaci di terza generazione come grazoprevir, glecaprevir, e voxilaprevir (file PDB 3sue, 6p6l, e 6nzt), che oggi sono largamente usati. Questi non si legano covalentemente all'enzima, ma riescono comunque ad aderire alla superficie della proteina perchè possiedono una struttura grande e ciclica che è in grado di inibire tutta una serie di proteasi mutate prodotte dal virus dell'epatite C. Spunti per ulteriori esplorazioni Negli archivi PDB vi sono molte strutture interessanti del dominio elicasi di HCV. Esplorate il file PDB 3kql per cominciare. Usate la pagina Ligand Page per esplorare le strutture di farmaci in maggior dettaglio. Per esempio provate a cercare grazoprevir.  Bibliografia BibliografiaHoughton, M. (2019) Hepatitis C virus: 30 years after its discovery. Cold Spring Harbor Persp Med 9: a037069 Ozen, A., Prachanronarong, K., Matthew, A. N., Soumana, D. I., Schiffer, C. A. (2019) Resistance outside the substrate envelope: hepatitis C NS3/4A protease inhibitors. Crit Rev Biochem Mol Biol 54: 11-26 Pol, S., Lagaye, S. (2019) The remarkable history of the hepatitis C virus. Genes Immun 20: 436-446 Gu, M., Rice, C. M. (2013) Structures of hepatitis C virus nonstructural protein required for replicase assembly and function. Curr Op Virol 3: 129-136 1cu1: Yao, N., Reichert, P., Taremi, S.S., Prosise, W.W., Weber, P.C. (1999) Molecular views of viral polyprotein processing revealed by the crystal structure of the hepatitis C virus bifunctional protease-helicase. Structure 7: 1353-1363 1r7g: Penin, F., Brass, V., Appel, N., Ramboarina, S., Montserret, R., Ficheux, D., Blum, H.E., Bartenschlager, R., Moradpour, D. (2004) Structure and function of the membrane anchor domain of hepatitis C virus nonstructural protein 5A. J Biol Chem 279: 40835-40843 1zh1: Tellinghuisen, T.L., Marcotrigiano, J., Rice, C.M. (2005) Structure of the zinc-binding domain of an essential component of the hepatitis C virus replicase. Nature 435: 374-379 2oc8: Prongay, A.J., Guo, Z., Yao, N., Pichardo, J., Fischmann, T., Strickland, C., Myers, J., Weber, P.C., Beyer, B.M., Ingram, R., Hong, Z., Prosise, W.W., Ramanathan, L., Taremi, S.S., Yarosh-Tomaine, T., Zhang, R., Senior, M., Yang, R.S., Malcolm, B., Arasappan, A., Bennett, F., Bogen, S.L., Chen, K., Jao, E., Liu, Y.T., Lovey, R.G., Saksena, A.K., Venkatraman, S., Girijavallabhan, V., Njoroge, F.G., Madison, V. (2007) Discovery of the HCV NS3/4A protease inhibitor (1R,5S)-N-[3-amino-1-(cyclobutylmethyl)-2,3-dioxopropyl]-3- [2(S)-[[[(1,1-dimethylethyl)amino]carbonyl]amino]-3,3-dimethyl-1-oxobutyl]- 6,6-dimethyl-3-azabicyclo[3.1.0]hexan-2(S)-carboxamide (Sch 503034) II. Key steps in structure-based optimization. J Med Chem 50: 2310-2318 3sue, 3sv6: Romano, K.P., Ali, A., Aydin, C., Soumana, D., Ozen, A., Deveau, L.M., Silver, C., Cao, H., Newton, A., Petropoulos, C.J., Huang, W., Schiffer, C.A. (2012) The molecular basis of drug resistance against hepatitis C virus NS3/4A protease inhibitors. PLoS Pathog 8: e1002832-e1002832 4wtg: Appleby, T.C., Perry, J.K., Murakami, E., Barauskas, O., Feng, J., Cho, A., Fox, D., Wetmore, D.R., McGrath, M.E., Ray, A.S., Sofia, M.J., Swaminathan, S., Edwards, T.E. (2015) Structural basis for RNA replication by the hepatitis C virus polymerase. Science 347: 771-775 6nzt: Taylor, J.G., Zipfel, S., Ramey, K., Vivian, R., Schrier, A., Karki, K.K., Katana, A., Kato, D., Kobayashi, T., Martinez, R., Sangi, M., Siegel, D., Tran, C.V., Yang, Z.Y., Zablocki, J., Yang, C.Y., Wang, Y., Wang, K., Chan, K., Barauskas, O., Cheng, G., Jin, D., Schultz, B.E., Appleby, T., Villasenor, A.G., Link, J.O. (2019) Discovery of the pan-genotypic hepatitis C virus NS3/4A protease inhibitor voxilaprevir (GS-9857): A component of Vosevi®. Bioorg Med Chem Lett 29: 2428-2436 6p6l: Timm, J., Kosovrasti, K., Henes, M., Leidner, F., Hou, S., Kurt-Yilmaz, N., Schiffer, C.A. (2020) Molecular and structural mechanism of pan-genotypic HCV NS3/4A protease inhibition by glecaprevir. ACS Chem Biol 15, 342-352
|
||||||
|
|
||||||
